import numpy as np
import pandas as pd
data = pd.Series([0.25, 0.5, 0.75, 1.0])
data7 Manipulación de datos con Pandas
Objetivo
El objetivo de esta clase es desarrollar habilidades en el manejo y análisis de datos utilizando pandas en Python, permitiendo a los estudiantes realizar operaciones de filtrado, agrupación y manipulación de datos para obtener información valiosa de conjuntos de datos reales.
7.1 Objeto Series de Pandas
Una Serie de Pandas es una matriz unidimensional de datos indexados. Se puede crear a partir de una lista o matriz de la siguiente manera:
Como vemos en la salida, la Serie envuelve una secuencia de valores y una secuencia de Índices, a los que podemos acceder con los atributos values e Index. Los values son simplemente una matriz NumPy:
data.valuesEl Index es un objeto tipo matriz de tipo pd.Index, que analizaremos con más detalle en breve.
data.indexAl igual que con una matriz NumPy, se puede acceder a los datos mediante el Index asociado a través de la conocida notación de corchetes de Python:
data[1]data[1:3]Sin embargo, como veremos, la Serie de Pandas es mucho más general y flexible que la matriz unidimensional NumPy que emula.
7.1.1 Series como matriz NumPy generalizada
De lo que hemos visto hasta ahora, puede parecer que el objeto Series es básicamente intercambiable con una matriz NumPy unidimensional. La diferencia esencial es la presencia del Index: mientras que la matriz Numpy tiene un Index entero implícitamente definido utilizado para acceder a los valores, el objeto Series de Pandas tiene un Index explícitamente definido asociado con los valores.
Esta definición explícita de Index le otorga al objeto Series capacidades adicionales. Por ejemplo, el Index no necesita ser un entero, sino que puede constar de valores de cualquier tipo deseado. Por ejemplo, si lo deseamos, podemos utilizar cadenas como Index:
data = pd.Series([0.25, 0.5, 0.75, 1.0],
index=['a', 'b', 'c', 'd'])
dataY el acceso al elemento funciona como se esperaba:
data['b']Incluso podemos utilizar Index no contiguos o no secuenciales:
data = pd.Series([0.25, 0.5, 0.75, 1.0],
index=[2, 5, 3, 7])
datadata[5]7.1.2 Series como diccionario especializado
De esta manera, puedes pensar en una Series de Pandas como si fuera una especialización de un diccionario de Python. Un diccionario es una estructura que asigna claves arbitrarias a un conjunto de valores arbitrarios y una Series es una estructura que asigna claves tipificadas a un conjunto de valores tipificados. Esta tipificación es importante: así como el código compilado específico de tipo detrás de una matriz NumPy la hace más eficiente que una lista de Python para ciertas operaciones, la información de tipo de una Series de Pandas la hace mucho más eficiente que los diccionarios de Python para ciertas operaciones.
La analogía de Series como diccionario se puede hacer aún más clara al construir un objeto Series directamente desde un diccionario de Python:
population_dict = {'California': 38332521,
'Texas': 26448193,
'New York': 19651127,
'Florida': 19552860,
'Illinois': 12882135}
population = pd.Series(population_dict)
populationDe forma predeterminada, se creará una Series donde el Index se extrae de las claves ordenadas. Desde aquí se puede realizar el acceso a elementos de estilo diccionario típico:
population['California']Sin embargo, a diferencia de un diccionario, la Series también admite operaciones de estilo matriz, como la segmentación:
population['California':'Texas']7.1.3 Construcción de objetos Series
Ya hemos visto algunas formas de construir una Series de Pandas desde cero; todas ellas son alguna versión de lo siguiente:
pd.Series(data, index=index)donde index es un argumento opcional y data puede ser una de muchas entidades.
Por ejemplo, data puede ser una lista o una matriz NumPy, en cuyo caso index tiene como valor predeterminado una secuencia de números enteros:
pd.Series([2, 4, 6])data puede ser un escalar, que se repite para llenar el index especificado:
pd.Series(5, index=[100, 200, 300])data puede ser un diccionario, en el que index tiene como valor predeterminado las claves ordenadas del diccionario:
pd.Series({2:'a', 1:'b', 3:'c'})En cada caso, el index se puede establecer explícitamente si se prefiere un resultado diferente:
pd.Series({2:'a', 1:'b', 3:'c'}, index=[3, 2])Tenga en cuenta que, en este caso, la Series se completa únicamente con las claves identificadas explícitamente.
7.2 El objeto DataFrame de Pandas
La siguiente estructura fundamental en Pandas es el DataFrame. Al igual que el objeto Series analizado en la sección anterior, el DataFrame puede considerarse como una generalización de una matriz NumPy o como una especialización de un diccionario de Python. Ahora analizaremos cada una de estas perspectivas.
7.2.1 DataFrame como una matriz NumPy generalizada
Si una serie es un análogo de una matriz unidimensional con índices flexibles, un DataFrame es un análogo de una matriz bidimensional con índices de fila flexibles y nombres de columna flexibles. Así como puedes pensar en una matriz bidimensional como una secuencia ordenada de columnas unidimensionales alineadas, puedes pensar en un DataFrame como una secuencia de objetos Series alineados. Aquí, por alineados queremos decir que comparten el mismo Index.
Para demostrar esto, construyamos primero una nueva Serie que enumere el área de cada uno de los cinco estados analizados en la sección anterior:
area_dict = {'California': 423967, 'Texas': 695662, 'New York': 141297,
'Florida': 170312, 'Illinois': 149995}
area = pd.Series(area_dict)
areaAhora que tenemos esto junto con la serie population de antes, podemos usar un diccionario para construir un solo objeto bidimensional que contenga esta información:
states = pd.DataFrame({'population': population,
'area': area})
statesAl igual que el objeto Series, el DataFrame tiene un atributo index que da acceso a las etiquetas de índice:
states.indexAdemás, el DataFrame tiene un atributo columns, que es un objeto Index que contiene las etiquetas de las columnas:
states.columnsPor lo tanto, el DataFrame puede considerarse como una generalización de una matriz NumPy bidimensional, donde tanto las filas como las columnas tienen un Index generalizado para acceder a los datos.
7.2.2 DataFrame como diccionario especializado
De manera similar, también podemos pensar en un DataFrame como una especialización de un diccionario. Donde un diccionario asigna una clave a un valor, un DataFrame asigna un nombre de columna a una Serie de datos de columna. Por ejemplo, al solicitar el atributo 'area' se devuelve el objeto Serie que contiene las áreas que vimos anteriormente:
states['area']Observe el posible punto de confusión aquí: en una matriz NumPy bidimensional, data[0] devolverá la primera fila. Para un DataFrame, data['col0'] devolverá la primera columna. Debido a esto, probablemente sea mejor pensar en los DataFrame como diccionarios generalizados en lugar de matrices generalizadas, aunque ambas formas de ver la situación pueden ser útiles.
7.2.3 Construcción de objetos DataFrame
Un DataFrame de Pandas se puede construir de distintas maneras. Aquí daremos varios ejemplos.
De un único objeto Series
Un DataFrame es una colección de objetos Series y un DataFrame de una sola columna se puede construir a partir de una sola Series:
pd.DataFrame(population, columns=['population'])De una lista de diccionarios
Cualquier lista de diccionarios se puede convertir en un DataFrame. Utilizaremos una lista de comprensión simple para crear algunos datos:
data = [{'x': i, '2*x': 2 * i, '2**x': 2**i}
for i in range(5)]
pd.DataFrame(data)Incluso si faltan algunas claves en el diccionario, Pandas las completará con valores NaN (es decir, no es un número):
pd.DataFrame([{'a': 1, 'b': 2}, {'b': 3, 'c': 4}])De un diccionario de objetos serie
Como vimos antes, un DataFrame también se puede construir a partir de un diccionario de objetos Series:
pd.DataFrame({'population': population,
'area': area})Desde una matriz NumPy bidimensional
Dada una matriz bidimensional de datos, podemos crear un DataFrame con cualquier nombre de columna e índice especificados. Si se omite, se utilizará un índice entero para cada uno:
pd.DataFrame(np.random.rand(3, 2),
columns=['foo', 'bar'],
index=['a', 'b', 'c'])7.3 Indexación y selección de datos
Ya concoemos los métodos y herramientas para acceder, establecer y modificar valores en matrices NumPy. Aquí veremos medios similares para acceder y modificar valores en los objetos Series y DataFrame de Pandas. Si ha utilizado los patrones NumPy, los patrones correspondientes en Pandas le resultarán muy familiares, aunque hay algunas peculiaridades que debe tener en cuenta.
Comenzaremos con el caso simple del objeto unidimensional Series y luego pasaremos al objeto bidimensional más complicado DataFrame.
7.3.1 Selección de datos en objetos Series
Como vimos en la sección anterior, un objeto Series actúa en un sentido como una matriz NumPy unidimensional y, en otro sentido, como un diccionario Python estándar. Si tenemos en cuenta estas dos analogías superpuestas, nos ayudará a comprender los patrones de indexación y selección de datos en estas matrices.
Serie como diccionario
Al igual que un diccionario, el objeto Series proporciona una asignación de una colección de claves a una colección de valores:
import pandas as pd
data = pd.Series([0.25, 0.5, 0.75, 1.0],
index=['a', 'b', 'c', 'd'])
datadata['b']También podemos utilizar expresiones y métodos de Python similares a diccionarios para examinar las claves/índices y valores:
'a' in datadata.keys()list(data.items())Los objetos Series pueden incluso modificarse con una sintaxis similar a la de un diccionario. Así como puedes ampliar un diccionario asignándolo a una nueva clave, puedes ampliar una Series asignándola a un nuevo valor de índice:
data['e'] = 1.25
dataEsta fácil mutabilidad de los objetos es una característica conveniente: en segundo plano, Pandas toma decisiones sobre el diseño de la memoria y la copia de datos que podría ser necesaria; el usuario generalmente no necesita preocuparse por estos problemas.
Serie como matriz unidimensional
Una Series se basa en esta interfaz tipo diccionario y proporciona una selección de elementos de estilo matriz a través de los mismos mecanismos básicos que las matrices NumPy, es decir, secciones, enmascaramiento e indexación elegante. Algunos ejemplos de estos son los siguientes:
# segmentación por índice explícito
data['a':'c']# corte por índice entero implícito
data[0:2]# enmascaramiento
data[(data > 0.3) & (data < 0.8)]# indexación elegante
data[['a', 'e']]Entre ellos, el corte puede ser la fuente de mayor confusión. Tenga en cuenta que al cortar con un índice explícito (es decir, data['a':'c']), el índice final se incluye en la porción, mientras que al cortar con un índice implícito (es decir, data[0:2]), el índice final se excluye de la porción.
Indexadores: loc e iloc
Estas convenciones de segmentación e indexación pueden ser una fuente de confusión. Por ejemplo, si su Series tiene un índice entero explícito, una operación de indexación como data[1] utilizará los índices explícitos, mientras que una operación de segmentación como data[1:3] utilizará el índice implícito de estilo Python.
data = pd.Series(['a', 'b', 'c'], index=[1, 3, 5])
data# índice explícito al indexar
data[1]# índice implícito al cortar
data[1:3]Debido a esta posible confusión en el caso de índices enteros, Pandas proporciona algunos atributos indexer especiales que exponen explícitamente ciertos esquemas de indexación. Estos no son métodos funcionales, sino atributos que exponen una interfaz de corte particular a los datos de la Series.
En primer lugar, el atributo loc permite la indexación y el corte que siempre hace referencia al índice explícito:
data.loc[1]data.loc[1:3]El atributo iloc permite la indexación y el corte que siempre hace referencia al índice implícito de estilo Python:
data.iloc[1]data.iloc[1:3]Un principio rector del código Python es que > Lo explícito es mejor que lo implícito
La naturaleza explícita de loc e iloc los hace muy útiles para mantener un código limpio y legible; especialmente en el caso de índices enteros, recomiendo utilizar ambos para hacer que el código sea más fácil de leer y entender y para evitar errores sutiles debido a la convención mixta de indexación/segmentación.
7.3.2 Selección de datos en DataFrame
Recuerde que un DataFrame actúa en muchos sentidos como una matriz bidimensional o estructurada, y en otros sentidos como un diccionario de estructuras Series que comparten el mismo índice. Puede resultar útil tener en cuenta estas analogías a medida que exploramos la selección de datos dentro de esta estructura.
DataFrame como diccionario
La primera analogía que consideraremos es el DataFrame como un diccionario de objetos Series relacionados. Volvamos a nuestro ejemplo de áreas y poblaciones de estados:
area = pd.Series({'California': 423967, 'Texas': 695662,
'New York': 141297, 'Florida': 170312,
'Illinois': 149995})
pop = pd.Series({'California': 38332521, 'Texas': 26448193,
'New York': 19651127, 'Florida': 19552860,
'Illinois': 12882135})
data = pd.DataFrame({'area':area, 'pop':pop})
dataSe puede acceder a las Series individuales que componen las columnas del DataFrame a través de la indexación de estilo diccionario del nombre de la columna:
data['area']De manera equivalente, podemos utilizar el acceso de estilo de atributo con nombres de columnas que son cadenas:
data.areaEste acceso a la columna de estilo de atributo en realidad accede exactamente al mismo objeto que el acceso de estilo de diccionario:
data.area is data['area']Si bien esta es una abreviatura útil, tenga en cuenta que no funciona en todos los casos. Por ejemplo, si los nombres de las columnas no son cadenas o si los nombres de las columnas entran en conflicto con los métodos del DataFrame, este acceso de estilo de atributo no es posible. Por ejemplo, el DataFrame tiene un método pop(), por lo que data.pop apuntará a esto en lugar de a la columna "pop":
data.pop is data['pop']En particular, debe evitar la tentación de intentar la asignación de columnas a través de atributos (es decir, utilizar data['pop'] = z en lugar de data.pop = z).
Al igual que con los objetos Series analizados anteriormente, esta sintaxis de estilo diccionario también se puede utilizar para modificar el objeto, en este caso agregando una nueva columna:
data['density'] = data['pop'] / data['area']
dataDataFrame como matriz bidimensional
Como se mencionó anteriormente, también podemos ver el DataFrame como una matriz bidimensional mejorada. Podemos examinar la matriz de datos subyacente sin procesar utilizando el atributo values:
data.valuesCon esta imagen en mente, se pueden realizar muchas observaciones familiares de tipo matriz en el propio DataFrame. Por ejemplo, podemos transponer el DataFrame completo para intercambiar filas y columnas:
data.TSin embargo, cuando se trata de la indexación de objetos DataFrame, está claro que la indexación de columnas en estilo diccionario impide nuestra capacidad de tratarlo simplemente como una matriz NumPy. En particular, al pasar un único índice a una matriz se accede a una fila:
data.values[0]y al pasar un único Index a un DataFrame se accede a una columna:
data['area']Por lo tanto, para la indexación de estilo matriz, necesitamos otra convención. Aquí Pandas vuelve a utilizar los indexadores loc e ilocmencionados anteriormente. Usando el indexador iloc, podemos indexar la matriz subyacente como si fuera una matriz NumPy simple (usando el índice implícito de estilo Python), pero el índice DataFrame y las etiquetas de columna se mantienen en el resultado:
data.iloc[:3, :2]De manera similar, utilizando el indexador loc podemos indexar los datos subyacentes en un estilo similar a una matriz pero utilizando los nombres de índice y columna explícitos:
data.loc[:'Illinois', :'pop']Se puede utilizar cualquiera de los patrones familiares de acceso a datos de estilo NumPy dentro de estos indexadores. Por ejemplo, en el indexador loc podemos combinar enmascaramiento e indexación elegante como en lo siguiente:
data.loc[data.density > 100, ['pop', 'density']]Cualquiera de estas convenciones de indexación también se puede utilizar para establecer o modificar valores; esto se hace de la manera estándar a la que probablemente esté acostumbrado al trabajar con NumPy:
data.iloc[0, 2] = 90
data7.3.3 Convenciones de indexación adicionales
Hay un par de convenciones de indexación adicionales que pueden parecer contrarias a la discusión anterior, pero sin embargo pueden resultar muy útiles en la práctica. En primer lugar, mientras que indexación se refiere a columnas, segmentación se refiere a filas:
data['Florida':'Illinois']Estas porciones también pueden hacer referencia a filas por número en lugar de por índice:
data[1:3]De manera similar, las operaciones de enmascaramiento directo también se interpretan por filas en lugar de por columnas:
data[data.density > 100]Estas dos convenciones son sintácticamente similares a aquellas de una matriz NumPy. Y si bien pueden no ajustarse exactamente al molde de las convenciones de Pandas, son bastante útiles en la práctica.
7.4 Operando con datos en Pandas
Una de las piezas esenciales de NumPy es la capacidad de realizar operaciones rápidas elemento por elemento, tanto con aritmética básica (suma, resta, multiplicación, etc.) como con operaciones más sofisticadas (funciones trigonométricas, funciones exponenciales y logarítmicas, etc.). Pandas hereda gran parte de esta funcionalidad de NumPy y las ufuncs son clave para esto.
Sin embargo, Pandas incluye un par de cambios útiles: para operaciones unarias como la negación y las funciones trigonométricas, estas ufuncs preservarán las etiquetas de índice y columna en la salida, y para operaciones binarias como la suma y la multiplicación, Pandas alineará automáticamente los índices al pasar los objetos a la ufunc. Esto significa que mantener el contexto de los datos y combinar datos de diferentes fuentes (ambas tareas potencialmente propensas a errores con matrices NumPy sin procesar) se vuelven esencialmente tareas infalibles con Pandas. Además, veremos que existen operaciones bien definidas entre estructuras unidimensionales tipo «Series» y estructuras bidimensionales tipo «DataFrame».
7.4.1 Ufuncs: Preservación de índice
Debido a que Pandas está diseñado para trabajar con NumPy, cualquier ufunc de NumPy funcionará en objetos Series y DataFrame de Pandas. Comencemos por definir una Serie y un DataFrame simples sobre los cuales demostrar esto:
import pandas as pd
import numpy as nprng = np.random.RandomState(42)
ser = pd.Series(rng.randint(0, 10, 4))
serdf = pd.DataFrame(rng.randint(0, 10, (3, 4)),
columns=['A', 'B', 'C', 'D'])
dfSi aplicamos una ufunc NumPy en cualquiera de estos objetos, el resultado será otro objeto Pandas con los índices conservados:
np.exp(ser)O, para un cálculo un poco más complejo:
np.sin(df * np.pi / 4)Cualquiera de las ufuncs discutidas se puede utilizar de manera similar.
7.4.2 UFuncs: Alineación de índice
Para operaciones binarias en dos objetos Series o DataFrame, Pandas alineará los índices en el proceso de realización de la operación. Esto es muy conveniente cuando trabajamos con datos incompletos, como veremos en algunos de los ejemplos que siguen.
Alineación de índices en la serie
A modo de ejemplo, supongamos que combinamos dos fuentes de datos diferentes y encontramos solo los tres principales estados de EE. UU. por área y los tres principales estados de EE. UU. por población:
area = pd.Series({'Alaska': 1723337, 'Texas': 695662,
'California': 423967}, name='area')
population = pd.Series({'California': 38332521, 'Texas': 26448193,
'New York': 19651127}, name='population')Veamos qué sucede cuando dividimos estos para calcular la densidad de población:
population / areaLa matriz resultante contiene la unión de los índices de las dos matrices de entrada.
Cualquier elemento para el cual uno u otro no tiene una entrada se marca con “NaN” o “No es un número”, que es la forma en que Pandas marca los datos faltantes. Esta coincidencia de índice se implementa de esta manera para cualquiera de las expresiones aritméticas integradas de Python; cualquier valor faltante se completa con NaN de manera predeterminada:
A = pd.Series([2, 4, 6], index=[0, 1, 2])
B = pd.Series([1, 3, 5], index=[1, 2, 3])
A + BSi el uso de valores NaN no es el comportamiento deseado, el valor de relleno se puede modificar utilizando métodos de objeto apropiados en lugar de los operadores. Por ejemplo, llamar a A.add(B) es equivalente a llamar a A + B, pero permite la especificación explícita opcional del valor de relleno para cualquier elemento en A o B que pueda faltar:
A.add(B, fill_value=0)Alineación de índices en DataFrame
Un tipo de alineación similar tiene lugar tanto para las columnas como para los índices cuando se realizan operaciones en DataFrame:
A = pd.DataFrame(rng.randint(0, 20, (2, 2)),
columns=list('AB'))
AB = pd.DataFrame(rng.randint(0, 10, (3, 3)),
columns=list('BAC'))
BA + BTenga en cuenta que los índices están alineados correctamente independientemente de su orden en los dos objetos, y los índices en el resultado están ordenados. Como fue el caso con Series, podemos usar el método aritmético del objeto asociado y pasar cualquier fill_value deseado para usar en lugar de las entradas faltantes. Aquí completaremos con la media de todos los valores en A (calculada apilando primero las filas de A):
fill = A.stack().mean()
A.add(B, fill_value=fill)7.4.3 Ufuncs: Operaciones entre DataFrame y Series
Al realizar operaciones entre un DataFrame y una Serie, la alineación del índice y de la columna se mantiene de manera similar. Las operaciones entre un DataFrame y una Serie son similares a las operaciones entre una matriz NumPy bidimensional y unidimensional. Consideremos una operación común, donde encontramos la diferencia de una matriz bidimensional y una de sus filas:
A = rng.randint(10, size=(3, 4))
AA - A[0]De acuerdo con las reglas de transmisión de NumPy, la resta entre una matriz bidimensional y una de sus filas se aplica fila por fila.
En Pandas, la convención funciona de manera similar por fila de manera predeterminada:
df = pd.DataFrame(A, columns=list('QRST'))
df - df.iloc[0]Si, en cambio, desea operar por columnas, puede utilizar los métodos de objeto mencionados anteriormente, mientras especifica la palabra clave axis:
df.subtract(df['R'], axis=0)Tenga en cuenta que estas operaciones DataFrame/Series, al igual que las operaciones analizadas anteriormente, alinearán automáticamente los índices entre los dos elementos:
halfrow = df.iloc[0, ::2]
halfrowdf - halfrowEsta preservación y alineación de índices y columnas significa que las operaciones sobre datos en Pandas siempre mantendrán el contexto de los datos, lo que evita los tipos de errores tontos que pueden surgir al trabajar con datos heterogéneos y/o desalineados en matrices NumPy sin procesar.
7.5 Manejo de datos faltantes
La diferencia entre los datos que se encuentran en muchos tutoriales y los datos del mundo real es que los datos del mundo real rara vez son limpios y homogéneos. En particular, en muchos conjuntos de datos interesantes faltará cierta cantidad de datos. Para complicar aún más las cosas, diferentes fuentes de datos pueden indicar datos faltantes de diferentes maneras.
En esta sección, analizaremos algunas consideraciones generales sobre los datos faltantes, analizaremos cómo Pandas elige representarlos y demostraremos algunas herramientas integradas de Pandas para manejar datos faltantes en Python. Aquí y a lo largo del libro, nos referiremos a los datos faltantes en general como valores nulos, NaN o NA.
7.5.1 Convenciones de datos faltantes
Hay una serie de esquemas que se han desarrollado para indicar la presencia de datos faltantes en una tabla o DataFrame. Generalmente, giran en torno a una de dos estrategias: utilizar una máscara que indica globalmente los valores faltantes o elegir un valor centinela que indica una entrada faltante.
En el enfoque de enmascaramiento, la máscara puede ser una matriz booleana completamente separada o puede implicar la apropiación de un bit en la representación de datos para indicar localmente el estado nulo de un valor.
En el enfoque centinela, el valor centinela podría ser alguna convención específica de los datos, como indicar un valor entero faltante con -9999 o algún patrón de bits raro, o podría ser una convención más global, como indicar un valor de punto flotante faltante con NaN (No es un número), un valor especial que es parte de la especificación de punto flotante IEEE.
Ninguno de estos enfoques está exento de desventajas: el uso de una matriz de máscaras independiente requiere la asignación de una matriz booleana adicional, lo que agrega sobrecarga tanto en almacenamiento como en computación. Un valor centinela reduce el rango de valores válidos que se pueden representar y puede requerir lógica adicional (a menudo no optimizada) en la aritmética de CPU y GPU. Los valores especiales comunes como NaN no están disponibles para todos los tipos de datos.
Como en la mayoría de los casos en los que no existe una opción universalmente óptima, los distintos idiomas y sistemas utilizan convenciones diferentes. Por ejemplo, el lenguaje R utiliza patrones de bits reservados dentro de cada tipo de datos como valores centinela que indican datos faltantes, mientras que el sistema SciDB utiliza un byte adicional adjunto a cada celda que indica un estado NA.
7.5.2 Datos faltantes en Pandas
La forma en que Pandas maneja los valores faltantes está limitada por su dependencia del paquete NumPy, que no tiene una noción incorporada de valores NA para tipos de datos que no sean de punto flotante.
Pandas podría haber seguido el ejemplo de R y especificar patrones de bits para cada tipo de datos individual para indicar la nulidad, pero este enfoque resulta bastante complicado de manejar. Si bien R contiene cuatro tipos de datos básicos, NumPy admite mucho más que esto: por ejemplo, mientras que R tiene un solo tipo de entero, NumPy admite catorce tipos de enteros básicos una vez que se tienen en cuenta las precisiones disponibles, el signo y el endianismo de la codificación. Reservar un patrón de bits específico en todos los tipos NumPy disponibles generaría una cantidad de sobrecarga inmanejable en la conversión especial de varias operaciones en casos especiales para varios tipos, lo que probablemente requeriría incluso una nueva bifurcación del paquete NumPy. Además, para los tipos de datos más pequeños (como los números enteros de 8 bits), sacrificar un bit para usarlo como máscara reducirá significativamente el rango de valores que puede representar.
NumPy tiene soporte para matrices enmascaradas, es decir, matrices que tienen una matriz de máscara booleana separada adjunta para marcar datos como “buenos” o “malos”. Pandas podría haber derivado de esto, pero la sobrecarga en almacenamiento, computación y mantenimiento del código hace que sea una opción poco atractiva.
Con estas restricciones en mente, Pandas eligió usar centinelas para los datos faltantes y además eligió usar dos valores nulos de Python ya existentes: el valor especial de punto flotante NaN y el objeto None de Python. Esta elección tiene algunos efectos secundarios, como veremos, pero en la práctica acaba siendo un buen compromiso en la mayoría de los casos de interés.
Ninguno: datos faltantes en Python
El primer valor centinela utilizado por Pandas es “Ninguno”, un objeto singleton de Python que a menudo se utiliza para datos faltantes en el código Python. Debido a que es un objeto Python, None no se puede usar en ninguna matriz NumPy/Pandas arbitraria, sino solo en matrices con tipo de datos 'object' (es decir, matrices de objetos Python):
import numpy as np
import pandas as pdvals1 = np.array([1, None, 3, 4])
vals1Este dtype=object significa que la mejor representación de tipo común que NumPy podría inferir para el contenido de la matriz es que son objetos de Python. Si bien este tipo de matriz de objetos es útil para algunos propósitos, cualquier operación sobre los datos se realizará a nivel de Python, con mucha más sobrecarga que las operaciones típicamente rápidas que se ven para matrices con tipos nativos:
for dtype in ['object', 'int']:
print("dtype =", dtype)
%timeit np.arange(1E6, dtype=dtype).sum()
print()El uso de objetos Python en una matriz también significa que si realiza agregaciones como sum() o min() en una matriz con un valor None, generalmente obtendrá un error:
#vals1.sum()Esto refleja el hecho de que la suma entre un entero y “Ninguno” no está definida.
NaN: Faltan datos numéricos
La otra representación de datos faltantes, «NaN» (acrónimo de Not a Number), es diferente; es un valor de punto flotante especial reconocido por todos los sistemas que utilizan la representación de punto flotante IEEE estándar:
vals2 = np.array([1, np.nan, 3, 4])
vals2.dtypeTenga en cuenta que NumPy eligió un tipo de punto flotante nativo para esta matriz: esto significa que, a diferencia de la matriz de objetos anterior, esta matriz admite operaciones rápidas introducidas en el código compilado. Debes tener en cuenta que «NaN» es un poco como un virus de datos: infecta cualquier otro objeto que toque. Independientemente de la operación, el resultado de la aritmética con NaN será otro NaN:
1 + np.nan0 * np.nanTenga en cuenta que esto significa que los agregados sobre los valores están bien definidos (es decir, no generan un error), pero no siempre son útiles:
vals2.sum(), vals2.min(), vals2.max()NumPy proporciona algunas agregaciones especiales que ignorarán estos valores faltantes:
np.nansum(vals2), np.nanmin(vals2), np.nanmax(vals2)Tenga en cuenta que “NaN” es específicamente un valor de punto flotante; no existe un valor NaN equivalente para números enteros, cadenas u otros tipos.
NaN y None en Pandas
Tanto “NaN” como “None” tienen su lugar, y Pandas está diseñado para manejar ambos de manera casi intercambiable, convirtiendo entre ellos cuando es apropiado:
pd.Series([1, np.nan, 2, None])Para los tipos que no tienen un valor centinela disponible, Pandas realiza una conversión de tipo automática cuando hay valores NA presentes. Por ejemplo, si establecemos un valor en una matriz de enteros en np.nan, se convertirá automáticamente a un tipo de punto flotante para acomodar el NA:
x = pd.Series(range(2), dtype=int)
xx[0] = None
xTenga en cuenta que, además de convertir la matriz de enteros a punto flotante, Pandas convierte automáticamente “Ninguno” en un valor “NaN”. (Tenga en cuenta que existe una propuesta para agregar un entero nativo NA a Pandas en el futuro; al momento de escribir este artículo, no se ha incluido).
Si bien este tipo de magia puede parecer un poco chapucera en comparación con el enfoque más unificado de los valores de NA en lenguajes de dominio específico como R, el enfoque de centinela/conversión de Pandas funciona bastante bien en la práctica y, en mi experiencia, rara vez causa problemas.
La siguiente tabla enumera las convenciones de conversión ascendente en Pandas cuando se introducen valores NA:
| Clase de tipo | Conversión al almacenar NA | Valor centinela de NA |
|---|---|---|
float |
Sin cambios | np.nan |
object |
Sin cambios | None o np.nan |
integer |
Convertir a float64 |
np.nan |
boolean |
Convertir a object |
None o np.nan |
Tenga en cuenta que en Pandas, los datos de cadena siempre se almacenan con un tipo de datos “objeto”.
7.5.3 Operando con valores nulos
Como hemos visto, Pandas trata a None y NaN como esencialmente intercambiables para indicar valores faltantes o nulos. Para facilitar esta convención, existen varios métodos útiles para detectar, eliminar y reemplazar valores nulos en las estructuras de datos de Pandas. Ellos son:
isnull(): Genera una máscara booleana que indica los valores faltantesnotnull(): Opuesto deisnull()dropna(): Devuelve una versión filtrada de los datosfillna(): Devuelve una copia de los datos con los valores faltantes completados o imputados
Concluiremos esta sección con una breve exploración y demostración de estas rutinas.
Detección de valores nulos
Las estructuras de datos de Pandas tienen dos métodos útiles para detectar datos nulos: isnull() y notnull(). Cualquiera de ellos devolverá una máscara booleana sobre los datos. Por ejemplo:
data = pd.Series([1, np.nan, 'hello', None])data.isnull()Las máscaras booleanas se pueden usar directamente como un índice Series o DataFrame:
data[data.notnull()]Los métodos isnull() y notnull() producen resultados booleanos similares para DataFrame.
Eliminación de valores nulos
Además del enmascaramiento utilizado anteriormente, existen los métodos de conveniencia, dropna() (que elimina los valores NA) y fillna() (que completa los valores NA). Para una Serie, El resultado es sencillo:
data.dropna()Para un “DataFrame”, hay más opciones. Considere el siguiente «DataFrame»:
df = pd.DataFrame([[1, np.nan, 2],
[2, 3, 5],
[np.nan, 4, 6]])
dfNo podemos eliminar valores individuales de un DataFrame; solo podemos eliminar filas o columnas completas. Dependiendo de la aplicación, es posible que desees uno u otro, por lo que dropna() ofrece varias opciones para un DataFrame.
De forma predeterminada, dropna() eliminará todas las filas en las que cualquier valor nulo esté presente:
df.dropna()Como alternativa, puede eliminar valores NA a lo largo de un eje diferente; axis=1 elimina todas las columnas que contienen un valor nulo:
df.dropna(axis='columns')Pero esto también deja caer algunos datos útiles; es posible que prefieras dejar caer filas o columnas con todos los valores NA, o una mayoría de los valores NA. Esto se puede especificar a través de los parámetros how o thresh, que permiten un control preciso de la cantidad de nulos que se permiten.
El valor predeterminado es how='any', de modo que cualquier fila o columna (dependiendo de la palabra clave axis) que contenga un valor nulo será descartada. También puede especificar how='all', que solo eliminará filas/columnas que sean todos valores nulos:
df[3] = np.nan
dfdf.dropna(axis='columns', how='all')Para un control más detallado, el parámetro thresh le permite especificar un número mínimo de valores no nulos que se conservarán en la fila/columna:
df.dropna(axis='rows', thresh=3)Aquí se han eliminado la primera y la última fila porque solo contienen dos valores no nulos.
Rellenar valores nulos
A veces, en lugar de eliminar los valores NA, es mejor reemplazarlos con un valor válido. Este valor podría ser un solo número como cero, o podría ser algún tipo de imputación o interpolación de los valores buenos. Puede hacer esto en el lugar usando el método isnull() como máscara, pero como es una operación tan común, Pandas proporciona el método fillna(), que devuelve una copia de la matriz con los valores nulos reemplazados.
Consideremos la siguiente «Serie»:
data = pd.Series([1, np.nan, 2, None, 3], index=list('abcde'))
dataPodemos llenar las entradas de NA con un solo valor, como cero:
data.fillna(0)Podemos especificar un relleno hacia adelante para propagar el valor anterior hacia adelante:
# relleno hacia adelante
data.ffill()O podemos especificar un relleno para propagar los siguientes valores hacia atrás:
# relleno
data.bfill()Para los “DataFrame”, las opciones son similares, pero también podemos especificar un “eje” a lo largo del cual se realizan los rellenos:
dfdf.ffill(axis=1)Tenga en cuenta que si un valor anterior no está disponible durante un relleno hacia adelante, el valor NA permanece.
7.6 Combinación de conjuntos de datos: concatenación y anexión
Algunos de los estudios de datos más interesantes surgen de la combinación de diferentes fuentes de datos. Estas operaciones pueden implicar cualquier cosa, desde una concatenación muy sencilla de dos conjuntos de datos diferentes hasta uniones y fusiones de estilo de base de datos más complicadas que manejan correctamente cualquier superposición entre los conjuntos de datos. Las «Series» y «DataFrame» se construyen con este tipo de operación en mente, y Pandas incluye funciones y métodos que hacen que este tipo de manejo de datos sea rápido y sencillo.
Aquí veremos la concatenación simple de Series y DataFrame con la función pd.concat; más adelante profundizaremos en fusiones y uniones en memoria más sofisticadas implementadas en Pandas.
Comenzamos con las importaciones estándar:
import pandas as pd
import numpy as npPara mayor comodidad, definiremos esta función que crea un DataFrame de un formato particular que será útil a continuación:
def make_df(cols, ind):
"""Quickly make a DataFrame"""
data = {c: [str(c) + str(i) for i in ind]
for c in cols}
return pd.DataFrame(data, ind)
# ejemplo DataFrame
make_df('ABC', range(3))Además, crearemos una clase rápida que nos permita mostrar varios DataFrame uno al lado del otro. El código hace uso del método especial _repr_html_, que IPython utiliza para implementar su visualización de objetos enriquecidos:
class display(object):
"""Display HTML representation of multiple objects"""
template = """<div style="float: left; padding: 10px;">
<p style='font-family:"Courier New", Courier, monospace'>{0}</p>{1}
</div>"""
def __init__(self, *args):
self.args = args
def _repr_html_(self):
return '\n'.join(self.template.format(a, eval(a)._repr_html_())
for a in self.args)
def __repr__(self):
return '\n\n'.join(a + '\n' + repr(eval(a))
for a in self.args)
El uso de esto quedará más claro a medida que continuemos nuestra discusión en la siguiente sección.
7.6.1 Concatenación simple con pd.concat
Pandas tiene una función, pd.concat(), que tiene una sintaxis similar a np.concatenate pero contiene una serie de opciones que discutiremos en breve:
# Signature in Pandas v0.18
pd.concat(objs, axis=0, join='outer', join_axes=None, ignore_index=False,
keys=None, levels=None, names=None, verify_integrity=False,
copy=True)pd.concat() se puede utilizar para una concatenación simple de objetos Series o DataFrame, al igual que np.concatenate() se puede utilizar para concatenaciones simples de matrices:
ser1 = pd.Series(['A', 'B', 'C'], index=[1, 2, 3])
ser2 = pd.Series(['D', 'E', 'F'], index=[4, 5, 6])
pd.concat([ser1, ser2])También funciona para concatenar objetos de dimensiones superiores, como “DataFrame”:
df1 = make_df('AB', [1, 2])
df2 = make_df('AB', [3, 4])
display('df1', 'df2', 'pd.concat([df1, df2])')De forma predeterminada, la concatenación se realiza fila por fila dentro del «DataFrame» (es decir, «axis=0»). Al igual que np.concatenate, pd.concat permite la especificación de un eje a lo largo del cual se llevará a cabo la concatenación. Consideremos el siguiente ejemplo:
df3 = make_df('AB', [0, 1])
df4 = make_df('CD', [0, 1])
display('df3', 'df4', "pd.concat([df3, df4], axis=1)")Podríamos haber especificado de manera equivalente axis=1; aquí hemos utilizado el más intuitivo axis='col'.
Indices duplicados
Una diferencia importante entre np.concatenate y pd.concat es que la concatenación de Pandas preserva los Indices, ¡incluso si el resultado tendrá Indices duplicados! Consideremos este sencillo ejemplo:
x = make_df('AB', [0, 1])
y = make_df('AB', [2, 3])
y.index = x.index # ¡Crea Indices duplicados!
display('x', 'y', 'pd.concat([x, y])')Observe los Indices repetidos en el resultado. Si bien esto es válido dentro de los “DataFrame”, el resultado suele ser indeseable. pd.concat() nos da algunas formas de manejarlo.
Detectar las repeticiones como un error
Si desea simplemente verificar que los Indices en el resultado de pd.concat() no se superpongan, puede especificar el indicador verify_integrity. Si se establece en Verdadero, la concatenación generará una excepción si hay Indices duplicados. A continuación se muestra un ejemplo en el que, para mayor claridad, capturaremos e imprimiremos el mensaje de error:
try:
pd.concat([x, y], verify_integrity=True)
except ValueError as e:
print("ValueError:", e)Ignorando el índice
A veces, el índice en sí no importa y preferirías simplemente ignorarlo. Esta opción se puede especificar utilizando el indicador ignore_index. Si se establece como verdadero, la concatenación creará un nuevo índice entero para la «Serie» resultante:
display('x', 'y', 'pd.concat([x, y], ignore_index=True)')7.6.2 Concatenación con uniones
En los ejemplos simples que acabamos de ver, estábamos principalmente concatenando DataFrame con nombres de columnas compartidos. En la práctica, los datos de diferentes fuentes pueden tener diferentes conjuntos de nombres de columnas, y pd.concat ofrece varias opciones en este caso. Considere la concatenación de los siguientes dos “DataFrame”, que tienen algunas (¡pero no todas!) columnas en común:
df5 = make_df('ABC', [1, 2])
df6 = make_df('BCD', [3, 4])
display('df5', 'df6', 'pd.concat([df5, df6])')De forma predeterminada, las entradas para las que no hay datos disponibles se rellenan con valores NA. Para cambiar esto, podemos especificar una de varias opciones para los parámetros join y join_axes de la función concatenar. De forma predeterminada, la unión es una unión de las columnas de entrada (join='outer'), pero podemos cambiar esto a una intersección de las columnas usando join='inner':
display('df5', 'df6',
"pd.concat([df5, df6], join='inner')")display('df1', 'df2', 'df1.append(df2)')Tenga en cuenta que, a diferencia de los métodos append() y extend() de las listas de Python, el método append() en Pandas no modifica el objeto original; en cambio, crea un nuevo objeto con los datos combinados. Tampoco es un método muy eficiente, porque implica la creación de un nuevo índice y un buffer de datos. Por lo tanto, si planea realizar múltiples operaciones de append, generalmente es mejor construir una lista de DataFrame y pasarlos todos a la vez a la función concat().
En la siguiente sección, veremos otro enfoque más poderoso para combinar datos de múltiples fuentes, las fusiones/uniones de estilo base de datos implementadas en pd.merge. Para obtener más información sobre concat(), append() y funcionalidades relacionadas, consulte “Merge, Join, and Concatenate” section de la documentación de Pandas.
7.7 Combinación de conjuntos de datos: fusionar y unir
Una característica esencial que ofrece Pandas son sus operaciones de unión y fusión en memoria de alto rendimiento. Si alguna vez ha trabajado con bases de datos, debería estar familiarizado con este tipo de interacción de datos. La interfaz principal para esto es la función pd.merge, y veremos algunos ejemplos de cómo puede funcionar en la práctica.
Para mayor comodidad, comenzaremos redefiniendo la funcionalidad display() de la sección anterior:
import pandas as pd
import numpy as np
class display(object):
"""Display HTML representation of multiple objects"""
template = """<div style="float: left; padding: 10px;">
<p style='font-family:"Courier New", Courier, monospace'>{0}</p>{1}
</div>"""
def __init__(self, *args):
self.args = args
def _repr_html_(self):
return '\n'.join(self.template.format(a, eval(a)._repr_html_())
for a in self.args)
def __repr__(self):
return '\n\n'.join(a + '\n' + repr(eval(a))
for a in self.args)7.7.1 Algebra relacional
El comportamiento implementado en pd.merge() es un subconjunto de lo que se conoce como álgebra relacional, que es un conjunto formal de reglas para manipular datos relacionales y forma la base conceptual de las operaciones disponibles en la mayoría de las bases de datos. La fortaleza del enfoque del álgebra relacional es que propone varias operaciones primitivas, que se convierten en los componentes básicos de operaciones más complicadas en cualquier conjunto de datos. Con este léxico de operaciones fundamentales implementado eficientemente en una base de datos u otro programa, se pueden realizar una amplia gama de operaciones compuestas bastante complicadas.
Pandas implementa varios de estos bloques de construcción fundamentales en la función pd.merge() y el método join() relacionado de Series y Dataframe. Como veremos, estos le permiten vincular de manera eficiente datos de diferentes fuentes.
7.7.2 Categorías de uniones
La función pd.merge() implementa varios tipos de uniones: uniones uno a uno, muchos a uno y muchos a muchos. Se accede a los tres tipos de uniones mediante una llamada idéntica a la interfaz pd.merge(); el tipo de unión realizada depende de la forma de los datos de entrada. Aquí mostraremos ejemplos simples de los tres tipos de fusiones y analizaremos opciones detalladas más adelante.
Uniones uno a uno
Quizás el tipo más simple de expresión de fusión es la unión uno a uno, que en muchos sentidos es muy similar a la concatenación por columnas. Como ejemplo concreto, considere los siguientes dos «DataFrames» que contienen información sobre varios empleados de una empresa:
df1 = pd.DataFrame({'employee': ['Bob', 'Jake', 'Lisa', 'Sue'],
'group': ['Accounting', 'Engineering', 'Engineering', 'HR']})
df2 = pd.DataFrame({'employee': ['Lisa', 'Bob', 'Jake', 'Sue'],
'hire_date': [2004, 2008, 2012, 2014]})
display('df1', 'df2')Para combinar esta información en un único DataFrame, podemos utilizar la función pd.merge():
df3 = pd.merge(df1, df2)
df3La función pd.merge() reconoce que cada DataFrame tiene una columna “empleado” y se une automáticamente utilizando esta columna como clave. El resultado de la fusión es un nuevo «DataFrame» que combina la información de las dos entradas. Tenga en cuenta que el orden de las entradas en cada columna no se mantiene necesariamente: en este caso, el orden de la columna “empleado” difiere entre df1 y df2, y la función pd.merge() lo tiene en cuenta correctamente. Además, tenga en cuenta que la fusión en general descarta el índice, excepto en el caso especial de fusiones por índice (consulte las palabras clave left_index y right_index, que se analizan en breve).
Uniones de varios a uno
Las uniones de varios a uno son uniones en las que una de las dos columnas clave contiene entradas duplicadas. En el caso de muchos a uno, el DataFrame resultante conservará esas entradas duplicadas según corresponda. Considere el siguiente ejemplo de una unión de varios a uno:
df4 = pd.DataFrame({'group': ['Accounting', 'Engineering', 'HR'],
'supervisor': ['Carly', 'Guido', 'Steve']})
display('df3', 'df4', 'pd.merge(df3, df4)')El DataFrame resultante tiene una columna adicional con la información del “supervisor”, donde la información se repite en una o más ubicaciones según lo requieran las entradas.
Uniones de muchos a muchos
Las uniones de muchos a muchos son un poco confusas conceptualmente, pero sin embargo están bien definidas. Si la columna clave tanto en la matriz izquierda como en la derecha contiene duplicados, el resultado es una fusión de muchos a muchos. Quizás esto quede más claro con un ejemplo concreto. Consideremos lo siguiente, donde tenemos un “DataFrame” que muestra una o más habilidades asociadas con un grupo en particular. Al realizar una unión de varios a varios, podemos recuperar las habilidades asociadas con cualquier persona individual:
df5 = pd.DataFrame({'group': ['Accounting', 'Accounting',
'Engineering', 'Engineering', 'HR', 'HR'],
'skills': ['math', 'spreadsheets', 'coding', 'linux',
'spreadsheets', 'organization']})
display('df1', 'df5', "pd.merge(df1, df5)")Estos tres tipos de uniones se pueden utilizar con otras herramientas Pandas para implementar una amplia gama de funcionalidades. Pero en la práctica, los conjuntos de datos rara vez son tan limpios como aquel con el que estamos trabajando aquí. En la siguiente sección, consideraremos algunas de las opciones proporcionadas por pd.merge() que le permiten ajustar el funcionamiento de las operaciones de unión.
7.7.3 Especificación de la clave de fusión
Ya hemos visto el comportamiento predeterminado de pd.merge(): busca uno o más nombres de columna coincidentes entre las dos entradas y los usa como clave. Sin embargo, a menudo los nombres de las columnas no coinciden tan bien y pd.merge() proporciona una variedad de opciones para manejar esto.
La palabra clave on
De forma más sencilla, puede especificar explícitamente el nombre de la columna clave utilizando la palabra clave on, que toma un nombre de columna o una lista de nombres de columnas:
display('df1', 'df2', "pd.merge(df1, df2, on='employee')")Esta opción solo funciona si tanto el DataFrame izquierdo como el derecho tienen el nombre de columna especificado.
Las palabras clave left_on y right_on
A veces, es posible que desees fusionar dos conjuntos de datos con diferentes nombres de columna; por ejemplo, podemos tener un conjunto de datos en el que el nombre del empleado esté etiquetado como “nombre” en lugar de “empleado”. En este caso, podemos usar las palabras clave left_on y right_on para especificar los dos nombres de columna:
df3 = pd.DataFrame({'name': ['Bob', 'Jake', 'Lisa', 'Sue'],
'salary': [70000, 80000, 120000, 90000]})
display('df1', 'df3', 'pd.merge(df1, df3, left_on="employee", right_on="name")')El resultado tiene una columna redundante que podemos eliminar si lo deseamos, por ejemplo, utilizando el método drop() de DataFrame:
pd.merge(df1, df3, left_on="employee", right_on="name").drop('name', axis=1)Las palabras clave left_index y right_index
A veces, en lugar de fusionar una columna, preferiría fusionar un índice. Por ejemplo, sus datos podrían verse así:
df1a = df1.set_index('employee')
df2a = df2.set_index('employee')
display('df1a', 'df2a')Puede utilizar el índice como clave para la fusión especificando los indicadores left_index y/o right_index en pd.merge():
display('df1a', 'df2a',
"pd.merge(df1a, df2a, left_index=True, right_index=True)")Para mayor comodidad, los DataFrame implementan el método join(), que realiza una fusión que, por defecto, une los Indices:
display('df1a', 'df2a', 'df1a.join(df2a)')Si desea mezclar Indices y columnas, puede combinar left_index con right_on o left_on con right_index para obtener el comportamiento deseado:
display('df1a', 'df3', "pd.merge(df1a, df3, left_index=True, right_on='name')")Todas estas opciones también funcionan con múltiples Indices y/o múltiples columnas; la interfaz para este comportamiento es muy intuitiva. Para obtener más información sobre esto, consulte “Merge, Join, and Concatenate” section de la documentación de Pandas.
7.7.4 Especificación de la aritmética de conjuntos para uniones
En todos los ejemplos anteriores hemos pasado por alto una consideración importante a la hora de realizar una unión: el tipo de aritmética de conjuntos utilizada en la unión. Esto ocurre cuando un valor aparece en una columna clave pero no en la otra. Considere este ejemplo:
df6 = pd.DataFrame({'name': ['Peter', 'Paul', 'Mary'],
'food': ['fish', 'beans', 'bread']},
columns=['name', 'food'])
df7 = pd.DataFrame({'name': ['Mary', 'Joseph'],
'drink': ['wine', 'beer']},
columns=['name', 'drink'])
display('df6', 'df7', 'pd.merge(df6, df7)')Aquí hemos fusionado dos conjuntos de datos que sólo tienen una única entrada de “nombre” en común: Mary. De forma predeterminada, el resultado contiene la intersección de los dos conjuntos de entradas; esto es lo que se conoce como unión interna. Podemos especificar esto explícitamente usando la palabra clave how, cuyo valor predeterminado es "inner":
pd.merge(df6, df7, how='inner')Otras opciones para la palabra clave cómo son 'exterior', 'izquierda' y 'derecha'. Una unión externa devuelve una unión sobre la unión de las columnas de entrada y completa todos los valores faltantes con NA:
display('df6', 'df7', "pd.merge(df6, df7, how='outer')")Las uniones izquierda y derecha devuelven uniones sobre las entradas izquierdas y derechas, respectivamente. Por ejemplo:
display('df6', 'df7', "pd.merge(df6, df7, how='left')")Las filas de salida ahora corresponden a las entradas en la entrada izquierda. how='right' funciona de manera similar.
Todas estas opciones se pueden aplicar directamente a cualquiera de los tipos de unión anteriores.
7.7.5 Nombres de columnas superpuestos: la palabra clave suffixes
Finalmente, puede terminar en un caso donde sus dos “DataFrame” de entrada tengan nombres de columna en conflicto. Considere este ejemplo:
df8 = pd.DataFrame({'name': ['Bob', 'Jake', 'Lisa', 'Sue'],
'rank': [1, 2, 3, 4]})
df9 = pd.DataFrame({'name': ['Bob', 'Jake', 'Lisa', 'Sue'],
'rank': [3, 1, 4, 2]})
display('df8', 'df9', 'pd.merge(df8, df9, on="name")')Debido a que la salida tendría dos nombres de columnas en conflicto, la función de combinación agrega automáticamente un sufijo _x o _y para que las columnas de salida sean únicas. Si estos valores predeterminados no son apropiados, es posible especificar un sufijo personalizado utilizando la palabra clave suffixes:
display('df8', 'df9', 'pd.merge(df8, df9, on="name", suffixes=["_L", "_R"])')Estos sufijos funcionan en cualquiera de los patrones de unión posibles y también funcionan si hay varias columnas superpuestas.
Para obtener más información sobre estos patrones, consulte Pandas “Merge, Join and Concatenate” documentation para obtener más información sobre estos temas.
7.7.6 Ejemplo
Las operaciones de fusión y unión surgen con mayor frecuencia cuando se combinan datos de diferentes fuentes. Aquí consideraremos un ejemplo de algunos datos sobre los estados de EE. UU. y sus poblaciones. Los archivos de datos se pueden encontrar en http://github.com/jakevdp/data-USstates/:
# A continuación se muestran los comandos de shell para descargar los datos.
!curl -O https://raw.githubusercontent.com/jakevdp/data-USstates/master/state-population.csv
!curl -O https://raw.githubusercontent.com/jakevdp/data-USstates/master/state-areas.csv
!curl -O https://raw.githubusercontent.com/jakevdp/data-USstates/master/state-abbrevs.csvEchemos un vistazo a los tres conjuntos de datos, utilizando la función read_csv() de Pandas:
pop = pd.read_csv('state-population.csv')
areas = pd.read_csv('state-areas.csv')
abbrevs = pd.read_csv('state-abbrevs.csv')
display('pop.head()', 'areas.head()', 'abbrevs.head()')Dada esta información, digamos que queremos calcular un resultado relativamente sencillo: clasificar los estados y territorios de EE. UU. según su densidad de población en 2010. Claramente tenemos los datos aquí para encontrar este resultado, pero tendremos que combinar los conjuntos de datos para encontrar el resultado.
Comenzaremos con una fusión de muchos a uno que nos dará el nombre del estado completo dentro de la población “DataFrame”. Queremos fusionar en función de la columna “estado/región” de “pop” y la columna “abreviatura” de “abbrevs”. Usaremos how='outer' para asegurarnos de que no se desperdicien datos debido a etiquetas no coincidentes.
merged = pd.merge(pop, abbrevs, how='outer',
left_on='state/region', right_on='abbreviation')
merged = merged.drop(labels='abbreviation', axis=1) # eliminar información duplicada
merged.head()Verifiquemos nuevamente si hubo alguna discrepancia aquí, lo que podemos hacer buscando filas con valores nulos:
merged.isnull().any()Parte de la información de “población” es nula; ¡averigüemos cuáles son!
merged[merged['population'].isnull()].head()Parece que todos los valores de población nula son de Puerto Rico antes del año 2000; esto probablemente se debe a que estos datos no están disponibles en la fuente original.
Más importante aún, vemos también que algunas de las nuevas entradas de estado también son nulas, lo que significa que no había una entrada correspondiente en la clave abbrevs. Veamos qué regiones carecen de esta coincidencia:
merged.loc[merged['state'].isnull(), 'state/region'].unique()Podemos inferir rápidamente el problema: nuestros datos de población incluyen entradas para Puerto Rico (PR) y Estados Unidos en su conjunto (USA), mientras que estas entradas no aparecen en la clave de abreviaturas de estados. Podemos solucionarlos rápidamente completando las entradas correspondientes:
merged.loc[merged['state/region'] == 'PR', 'state'] = 'Puerto Rico'
merged.loc[merged['state/region'] == 'USA', 'state'] = 'United States'
merged.isnull().any()No más valores nulos en la columna estado: ¡estamos listos!
Ahora podemos fusionar el resultado con los datos del área utilizando un procedimiento similar. Al examinar nuestros resultados, querremos unirnos en la columna “estado” en ambos:
final = pd.merge(merged, areas, on='state', how='left')
final.head()Nuevamente, verifiquemos si hay valores nulos para ver si hubo alguna discrepancia:
final.isnull().any()Hay valores nulos en la columna área; podemos echar un vistazo para ver qué regiones se ignoraron aquí:
final['state'][final['area (sq. mi)'].isnull()].unique()Vemos que nuestro DataFrame de áreas no contiene el área de los Estados Unidos en su totalidad. Podríamos insertar el valor apropiado (usando la suma de todas las áreas estatales, por ejemplo), pero en este caso simplemente eliminaremos los valores nulos porque la densidad de población de todos los Estados Unidos no es relevante para nuestra discusión actual:
final.dropna(inplace=True)
final.head()Ahora tenemos todos los datos que necesitamos. Para responder a la pregunta de interés, primero seleccionemos la parte de los datos correspondiente al año 2010 y a la población total. Usaremos la función query() para hacer esto rápidamente (esto requiere que el paquete numexpr esté instalado):
data2010 = final.query("year == 2010 & ages == 'total'")
data2010.head()Ahora calculemos la densidad de población y mostrémosla en orden. Comenzaremos reindexando nuestros datos sobre el estado y luego calcularemos el resultado:
data2010.set_index('state', inplace=True)
density = data2010['population'] / data2010['area (sq. mi)']density.sort_values(ascending=False, inplace=True)
density.head()El resultado es una clasificación de los estados de EE. UU. más Washington, DC y Puerto Rico en orden de su densidad de población en 2010, en residentes por milla cuadrada. Podemos ver que, con diferencia, la región más densa en este conjunto de datos es Washington, DC (es decir, el Distrito de Columbia); entre los estados, el más denso es Nueva Jersey.
También podemos consultar el final de la lista:
density.tail()Vemos que el estado menos denso, con diferencia, es Alaska, con un promedio de poco más de un residente por milla cuadrada.
Este tipo de fusión desordenada de datos es una tarea común cuando se intenta responder preguntas utilizando fuentes de datos del mundo real. ¡Espero que este ejemplo te haya dado una idea de las formas en que puedes combinar las herramientas que hemos cubierto para obtener información de tus datos!
7.8 Agregación y agrupación
Una parte esencial del análisis de datos grandes es el resumen eficiente: calcular agregaciones como sum(), mean(), median(), min() y max(), en las que un solo número brinda información sobre la naturaleza de un conjunto de datos potencialmente grande. En esta sección, exploraremos las agregaciones en Pandas, desde operaciones simples similares a las que hemos visto en las matrices NumPy, hasta operaciones más sofisticadas basadas en el concepto de “groupby”.
Para mayor comodidad, utilizaremos la misma función mágica display que hemos visto en secciones anteriores:
import numpy as np
import pandas as pd
class display(object):
"""Display HTML representation of multiple objects"""
template = """<div style="float: left; padding: 10px;">
<p style='font-family:"Courier New", Courier, monospace'>{0}</p>{1}
</div>"""
def __init__(self, *args):
self.args = args
def _repr_html_(self):
return '\n'.join(self.template.format(a, eval(a)._repr_html_())
for a in self.args)
def __repr__(self):
return '\n\n'.join(a + '\n' + repr(eval(a))
for a in self.args)7.8.1 Datos de los planetas
Aquí utilizaremos el conjunto de datos Planetas, disponible a través del paquete Seaborn. Proporciona información sobre planetas que los astrónomos han descubierto alrededor de otras estrellas (conocidos como planetas extrasolares o exoplanetas para abreviar). Se puede descargar con un simple comando Seaborn:
import seaborn as sns
planets = sns.load_dataset('planets')
planets.shapeplanets.head()Aquí encontrará algunos detalles sobre los más de 1.000 planetas extrasolares descubiertos hasta 2014.
7.8.2 Agregación simple en Pandas
Anteriormente, exploramos algunas de las agregaciones de datos disponibles para las matrices NumPy. Al igual que con una matriz NumPy unidimensional, para una «Serie» de Pandas los agregados devuelven un único valor:
rng = np.random.RandomState(42)
ser = pd.Series(rng.rand(5))
serser.sum()ser.mean()Para un DataFrame, de forma predeterminada los agregados devuelven resultados dentro de cada columna:
df = pd.DataFrame({'A': rng.rand(5),
'B': rng.rand(5)})
dfdf.mean()Al especificar el argumento axis, puedes agregar dentro de cada fila:
df.mean(axis='columns')Las Series y los DataFrame de Pandas incluyen todos los agregados comunes ; además, hay un método conveniente describe() que calcula varios agregados comunes para cada columna y devuelve el resultado. Usemos esto en los datos de Planetas, por ahora descartando filas con valores faltantes:
planets.dropna().describe()Esta puede ser una forma útil de comenzar a comprender las propiedades generales de un conjunto de datos. Por ejemplo, vemos en la columna “año” que, si bien los exoplanetas se descubrieron ya en 1989, la mitad de todos los exoplanetas conocidos no se descubrieron hasta 2010 o después. Esto se debe en gran medida a la misión Kepler, que es un telescopio espacial diseñado específicamente para encontrar planetas eclipsantes alrededor de otras estrellas.
La siguiente tabla resume algunas otras agregaciones integradas de Pandas:
| Agregación | Descripción |
|---|---|
count() |
Número total de elementos |
first(), last() |
Primer y último elemento |
mean(), median() |
Media y mediana |
min(), max() |
Mínimo y máximo |
std(), var() |
Desviación estándar y varianza |
mad() |
Desviación absoluta media |
prod() |
Producto de todos los artículos |
sum() |
Suma de todos los elementos |
Todos estos son métodos de objetos DataFrame y Series.
Sin embargo, para profundizar en los datos, a menudo los agregados simples no son suficientes. El siguiente nivel de resumen de datos es la operación “groupby”, que permite calcular de forma rápida y eficiente agregados en subconjuntos de datos.
7.8.3 GroupBy
Las agregaciones simples pueden darle una idea de su conjunto de datos, pero a menudo preferiríamos agregar condicionalmente en alguna etiqueta o índice: esto se implementa en la llamada operación “groupby”. El nombre “agrupar por” proviene de un comando en el lenguaje de base de datos SQL, pero tal vez sea más esclarecedor pensar en él en los términos acuñados por primera vez por Hadley Wickham, de Rstats: dividir, aplicar, combinar.
Dividir, aplicar, combinar
Un ejemplo canónico de esta operación de dividir-aplicar-combinar, donde Apply es una agregación de suma, se ilustra en esta figura:
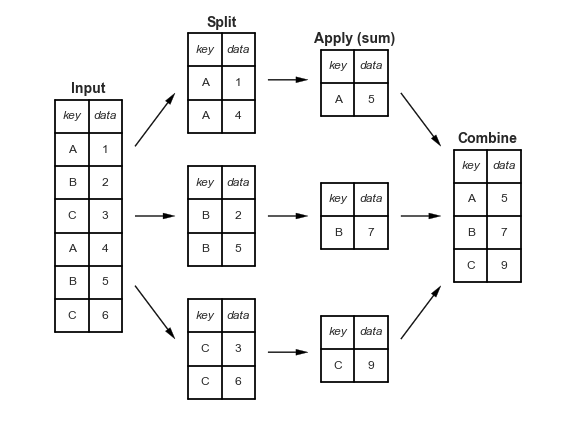
Esto deja claro lo que logra el groupby:
- El paso dividir implica dividir y agrupar un
DataFramedependiendo del valor de la clave especificada. - El paso aplicar implica calcular alguna función, generalmente un agregado, una transformación o un filtrado, dentro de los grupos individuales.
- El paso combinar fusiona los resultados de estas operaciones en una matriz de salida.
Si bien esto podría hacerse de manera manual utilizando alguna combinación de los comandos de enmascaramiento, agregación y fusión que se mencionaron anteriormente, es importante tener en cuenta que las divisiones intermedias no necesitan instanciarse explícitamente. En cambio, el GroupBy puede (a menudo) hacer esto en una sola pasada sobre los datos, actualizando la suma, la media, el recuento, el mínimo u otro agregado para cada grupo a lo largo del camino. El poder de GroupBy es que abstrae estos pasos: el usuario no necesita pensar en cómo se realiza el cálculo bajo el capó, sino que piensa en la operación como un todo.
Como ejemplo concreto, veamos el uso de Pandas para el cálculo que se muestra en este diagrama. Comenzaremos creando la entrada DataFrame:
df = pd.DataFrame({'key': ['A', 'B', 'C', 'A', 'B', 'C'],
'data': range(6)}, columns=['key', 'data'])
dfLa operación más básica de dividir-aplicar-combinar se puede calcular con el método groupby() de DataFrame, pasando el nombre de la columna clave deseada:
df.groupby('key')Tenga en cuenta que lo que se devuelve no es un conjunto de DataFrame, sino un objeto DataFrameGroupBy. Este objeto es donde está la magia: puedes pensar en él como una vista especial del «DataFrame», que está preparado para profundizar en los grupos pero no realiza ningún cálculo real hasta que se aplica la agregación. Este enfoque de “evaluación perezosa” significa que los agregados comunes se pueden implementar de manera muy eficiente y de una manera casi transparente para el usuario.
Para producir un resultado, podemos aplicar un agregado a este objeto DataFrameGroupBy, que realizará los pasos de aplicación/combinación adecuados para producir el resultado deseado:
df.groupby('key').sum()El método sum() es sólo una posibilidad aquí; puede aplicar virtualmente cualquier función de agregación común de Pandas o NumPy, así como virtualmente cualquier operación DataFrame válida, como veremos en la siguiente discusión.
El objeto GroupBy
El objeto GroupBy es una abstracción muy flexible. En muchos sentidos, puedes tratarlo como si fuera una colección de “DataFrame” y hace las cosas difíciles por dentro. Veamos algunos ejemplos con los datos de Planets.
Quizás las operaciones más importantes que permite un GroupBy son agregar, filtrar, transformar y aplicar.
Indexación de columnas
El objeto GroupBy admite la indexación de columnas de la misma manera que DataFrame y devuelve un objeto GroupBy modificado. Por ejemplo:
planets.groupby('method')planets.groupby('method')['orbital_period']Aquí hemos seleccionado un grupo «Series» particular del grupo «DataFrame» original por referencia a su nombre de columna. Al igual que con el objeto GroupBy, no se realiza ningún cálculo hasta que llamamos a algún agregado en el objeto:
planets.groupby('method')['orbital_period'].median()Esto da una idea de la escala general de períodos orbitales (en días) a los que cada método es sensible.
Iteración sobre grupos
El objeto GroupBy admite la iteración directa sobre los grupos y devuelve cada grupo como una Serie o DataFrame:
for (method, group) in planets.groupby('method'):
print("{0:30s} shape={1}".format(method, group.shape))Esto puede ser útil para hacer ciertas cosas manualmente, aunque a menudo es mucho más rápido utilizar la funcionalidad incorporada “aplicar”, que analizaremos en breve.
Métodos de envío
Mediante cierta magia de clase de Python, cualquier método no implementado explícitamente por el objeto GroupBy será pasado y llamado en los grupos, ya sean objetos DataFrame o Series. Por ejemplo, puede utilizar el método describe() de DataFrame para realizar un conjunto de agregaciones que describan cada grupo en los datos:
planets.groupby('method')['year'].describe().unstack()Mirar esta tabla nos ayuda a comprender mejor los datos: por ejemplo, la gran mayoría de los planetas han sido descubiertos mediante los métodos de velocidad radial y de tránsito, aunque estos últimos solo se volvieron comunes (debido a telescopios nuevos y más precisos) en la última década. Los métodos más nuevos parecen ser la variación del tiempo de tránsito y la modulación del brillo orbital, que no se utilizaron para descubrir un nuevo planeta hasta 2011.
Este es sólo un ejemplo de la utilidad de los métodos de despacho. Tenga en cuenta que se aplican a cada grupo individual y luego los resultados se combinan dentro de GroupBy y se devuelven. Nuevamente, cualquier método DataFrame/Series válido se puede usar en el objeto GroupBy correspondiente, lo que permite algunas operaciones muy flexibles y poderosas.
Agregar, filtrar, transformar, aplicar
La discusión anterior se centró en la agregación para la operación de combinación, pero hay más opciones disponibles. En particular, los objetos GroupBy tienen métodos agregate(), filter(), transform() y apply() que implementan eficientemente una variedad de operaciones útiles antes de combinar los datos agrupados.
Para los fines de las siguientes subsecciones, utilizaremos este DataFrame:
rng = np.random.RandomState(0)
df = pd.DataFrame({'key': ['A', 'B', 'C', 'A', 'B', 'C'],
'data1': range(6),
'data2': rng.randint(0, 10, 6)},
columns = ['key', 'data1', 'data2'])
dfAgregación
Ahora estamos familiarizados con las agregaciones GroupBy con sum(), median() y similares, pero el método agregate() permite aún más flexibilidad. Puede tomar una cadena, una función o una lista de ellas y calcular todos los agregados a la vez. He aquí un ejemplo rápido que combina todo esto:
df.groupby('key').aggregate(['min', 'median', 'max'])Otro patrón útil es pasar un diccionario que asigne los nombres de las columnas a las operaciones que se aplicarán en esa columna:
df.groupby('key').aggregate({'data1': 'min',
'data2': 'max'})Filtrado
Una operación de filtrado le permite eliminar datos según las propiedades del grupo. Por ejemplo, podríamos querer conservar todos los grupos en los que la desviación estándar sea mayor que algún valor crítico:
def filter_func(x):
return x['data2'].std() > 4
display('df', "df.groupby('key').std()", "df.groupby('key').filter(filter_func)")La función de filtro debe devolver un valor booleano que especifique si el grupo pasa el filtro. En este caso, como el grupo A no tiene una desviación estándar mayor que 4, se lo excluye del resultado.
Transformación
Mientras que la agregación debe devolver una versión reducida de los datos, la transformación puede devolver una versión transformada de los datos completos para recombinarlos. Para tal transformación, la salida tiene la misma forma que la entrada. Un ejemplo común es centrar los datos restando la media del grupo:
df.groupby('key').transform(lambda x: x - x.mean())El método apply()
El método apply() le permite aplicar una función arbitraria a los resultados del grupo. La función debe tomar un DataFrame y devolver un objeto Pandas (por ejemplo, DataFrame, Series) o un escalar; la operación de combinación se adaptará al tipo de salida devuelta.
Por ejemplo, aquí hay un apply() que normaliza la primera columna por la suma de la segunda:
def norm_by_data2(x):
# x es un DataFrame de valores de grupo
x['data1'] /= x['data2'].sum()
return x
display('df', "df.groupby('key').apply(norm_by_data2)")apply() dentro de un GroupBy es bastante flexible: el único criterio es que la función toma un DataFrame y devuelve un objeto Pandas o un escalar; ¡lo que haga en el medio depende de usted!
Especificación de la clave dividida
En los ejemplos simples presentados anteriormente, dividimos el “DataFrame” en un solo nombre de columna. Esta es sólo una de las muchas opciones mediante las cuales se pueden definir los grupos; aquí repasaremos algunas otras opciones para la especificación de grupos.
Una lista, matriz, serie o índice que proporciona las claves de agrupación
La clave puede ser cualquier serie o lista con una longitud que coincida con la del DataFrame. Por ejemplo:
L = [0, 1, 0, 1, 2, 0]
display('df', 'df.groupby(L).sum()')Por supuesto, esto significa que hay otra forma más detallada de lograr el df.groupby('key') de antes:
display('df', "df.groupby(df['key']).sum()")Un diccionario o serie que asigna el índice al grupo
Otro método es proporcionar un diccionario que asigne valores de índice a las claves del grupo:
df2 = df.set_index('key')
mapping = {'A': 'vowel', 'B': 'consonant', 'C': 'consonant'}
display('df2', 'df2.groupby(mapping).sum()')Cualquier función de Python
De manera similar al mapeo, puedes pasar cualquier función de Python que ingrese el valor del índice y genere el grupo:
display('df2', 'df2.groupby(str.lower).mean()')Una lista de claves válidas
Además, cualquiera de las opciones clave anteriores se pueden combinar para agruparlas en un índice múltiple:
df2.groupby([str.lower, mapping]).mean()Ejemplo de agrupación
Como ejemplo de esto, en un par de líneas de código Python podemos juntar todo esto y contar los planetas descubiertos por método y por década:
decade = 10 * (planets['year'] // 10)
decade = decade.astype(str) + 's'
decade.name = 'decade'
planets.groupby(['method', decade])['number'].sum().unstack().fillna(0)Esto demuestra el poder de combinar muchas de las operaciones que hemos discutido hasta este punto cuando observamos conjuntos de datos realistas. ¡Obtenemos inmediatamente una comprensión aproximada de cuándo y cómo se descubrieron los planetas a lo largo de las últimas décadas!
Aquí sugeriría profundizar en estas pocas líneas de código y evaluar los pasos individuales para asegurarse de comprender exactamente lo que están haciendo con el resultado. Ciertamente es un ejemplo algo complicado, pero comprender estas piezas le brindará los medios para explorar de manera similar sus propios datos.
7.9 Ejercicios prácticos
- Cree un nuevo Notebook.
- Guarde el archivo como Ejercicios_practicos_clase_7.ipynb.
- Asigne un título H1 con su nombre.
7.9.1 Ejercicio práctico 1
Se tiene el archivo ventas.csv con las ventas de una tienda en los últimos seis meses. El archivo contiene las siguientes columnas: Fecha, Producto, Cantidad, PrecioUnitario, Categoria.
- Cargar el archivo CSV en un DataFrame.
- Mostrar las primeras 5 filas del DataFrame para familiarizarse con los datos.
- Calcular el total de ingresos (Cantidad * PrecioUnitario) y añadirlo como una nueva columna.
- Filtrar y mostrar las ventas realizadas en el mes de junio.
- Agrupar las ventas por Categoria y calcular el ingreso total por cada categoría.
- Identificar los productos con mayores ventas en términos de cantidad y de ingresos.
7.9.2 Ejercicio práctico 2
Se tiene el archivo Excel empleados.csv con la información de empleados de una empresa. El archivo contiene las siguientes columnas: ID, Nombre, Departamento, Salario, Edad, AñosEnLaEmpresa.
- Cargar el archivo Excel en un DataFrame.
- Mostrar las columnas Nombre, Salario y Departamento de todos los empleados.
- Calcular el salario promedio de los empleados por departamento.
- Filtrar los empleados que tienen más de 10 años en la empresa y un salario mayor a $3.500.000.
- Añadir una columna que calcule el bono de fin de año (10% del salario si tiene más de 5 años en la empresa, 5% en caso contrario).
- Ordenar el DataFrame por Salario de mayor a menor y mostrar los 5 empleados con mayor salario.